
摘要:通过计算机辅助药物设计策略及固相合成法设计并筛选具有全新结构的αvβ6多肽配体,并用酶联免疫吸附法(ELISA)测定多肽配体与αvβ6的结合亲和力,建立αvβ6多肽配体的筛选方案.首 先 用 Sybyl-X1.3 对αvβ6 多 肽 配 体 及 天 然 配 体 进 行 分 子 对 接;其 次 用Amber16对分子对接结果进行分子动力学模拟,确定多肽配体与αvβ6蛋白之间的结合模式,并以 RGDLXXL(X为任意氨基酸)为多肽配体核心结构,通过逐步延伸氨基酸构建虚拟肽库,筛选获得长度为7~10个氨基酸的多肽配体,进一步通过相似氨基酸替换的方法设计筛选不同于 RGDLXXL核心的新的多肽配体;最后用固相合成法合成新设计的多肽配体,利用间接 ELISA 法测定多肽配体-αvβ6的结合亲和力.已有多肽配体和天然配体的分子对接以及分子动力学模拟结果表明,αvβ6与配体的结合主要通过 Asp218和多肽配体之间形成氢键,以及 Mg2+ 和多肽配体形成金属螯合作用完成.结合虚拟组合筛选与相似氨基酸替换,发现GRTDLGTLLFR,GRRTDLATIHG,RTDVGRVRGRG 和 RGDVGRVGR 等多肽均满足该结合模式,RTDVGRVRGRG 与αvβ6的亲和力为10.76μmol/L.可见 RTDVGRVRGRG与αvβ6亲和力良好,是一条全新的αvβ6多肽配体.
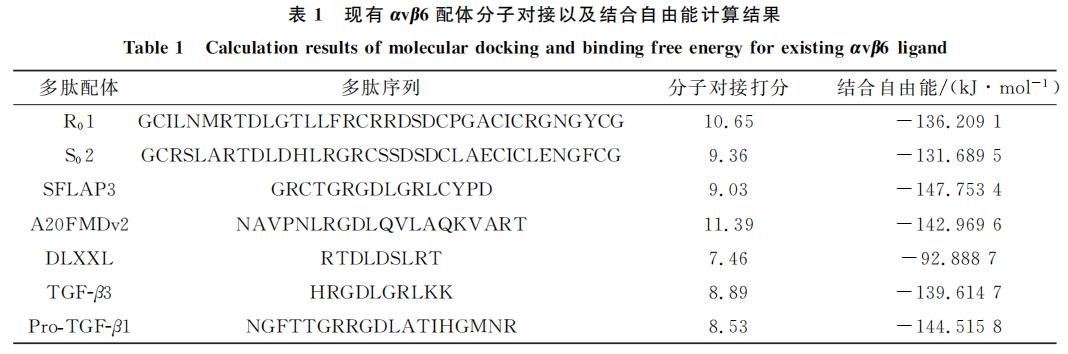
目前文献报道的靶向αvβ6配体包括小分子配体和多肽类配体,其中多肽类配体主要包括定向工程化噬菌体展示获得的多肽 R01和 S02[4],基于向日葵胰蛋白酶抑制剂噬菌体展示筛选得到的多肽SFLAP3[5],源于口蹄疫病毒外壳的多肽 A20FMDv2[6],噬菌体展示文库筛选得到的多肽 DLXXL[7],以及天然配体 Pro-TGF-β1和 TGF-β3[8].其中[18F]氟苯甲酰基标记的肽[18F]FBA-A20FMDv2已在人类临床试验中用作 PET 放射性示踪剂,用于特发性肺纤维化的诊断和治疗评估.研究表明,尽管多肽配体没有直接的抗癌活性,但可以作为分子成像载体用于癌症等疾病诊断,以及通过与药物或纳米粒的偶联进行整合素靶向肿瘤治疗.靶向整合素放射药物已是比较成熟的研究方向,此类药物结合了两种关键元素:一种靶向化合物/配体和一种放射性同位素[9].靶向放射性药物可以与肿瘤组织中的特异性靶点结合并聚集,通过放射性射线产生电离辐射作用,由于正常组织细胞与肿瘤组织细胞对射线的敏感性 不 同,因 此 可 以 实 现 肿 瘤 的 靶 向 治 疗 或 根 据 放 射 性 射 线 对 肿 瘤 进 行 精 准 诊 断.如177Lu-PSMA-617(Novartis)即将被批准用于前列腺癌的诊疗[10],由北京大学王凡课题组研发的中国首个核医学肿瘤显像诊断1类新药99mTc-3PRGD2已完成三期临床[11].因此开发具有靶向αvβ6作用的多肽配体具有重要意义.
针对目前αvβ6结合多肽基本含有 RGDLXXL(X 为任意氨基酸)核心结构,且该结构处于专利保护范围内,本文拟筛选设计非专利范围内全新结构的αvβ6结合多肽配体.先通过分子模拟方法分析αvβ6与多肽配体的结合模式,再通过计算机辅助药物设计方法发现一种全新结构的αvβ6多肽配体,该配体具有良好的结合亲和力,可为后续的靶向αvβ6多肽和多肽-核素复合物研究提供理论与实际研究指导,是一个具有发展前景的先导化合物.
1 材料与方法
Fmoc-氨基酸(上海麦克林生化科技股份有限公司),2-氯代三苯甲基氯-树脂(阿拉丁试剂(上海)有限公司),二氯甲烷(DCM)、二甲基甲酰胺(DMF)、二异丙基乙胺(DIEA)和乙腈等(国药集团化学试剂有 限 公 司),人 整 合 素 αvβ6 蛋 白 (MedChemExpressLLC)、链 霉 亲 和 素-辣 根 过 氧 化 物 酶(Streptavidin-HPR)和 3,3′,5,5′-四 甲 基 联 苯 胺 (TMB)显 色 液 (上 海 碧 云 天 生 物 技A20FMDv2(术 有 限 公 司),NAVPNLRGDLQVLAQKVART)、A20FMDv2-Biotin(Biotin-NAVPNLRGDLQVLAQKVART)和 A20FMDv2-GRD(NAVPNLGRDLQVLAQKVART)(核欣(苏州)医药科技有限公司),酶联免疫吸附试剂盒(江苏酶免实业有限公司).
液相制备色谱仪(LC-20AP 型,日本岛津公司);三重四级杆液-质联用仪(AGILENT6470 型,美国安捷伦科技有限公司);全波长酶标仪(MultiskanSkyHigh型,美国赛默飞世尔科技公司).
采用 Sybyl-X1.3 进 行 分 子 对 接,靶 蛋 白αvβ6 源 自 蛋 白 质 结 构 数 据 库 (Protein Data Bank,http://www.rcsb.org,ID:5FFO),配 体 来 源 于 文 献、有 机 小 分 子 生 物 活 性 数 据 库 (PubChem,https://pubchem.ncbi.nlm.nih.gov)和药物化学数据库(Chembl,https://www.ebi.ac.uk/chembl/)等.使用Sybyl软件的Surflex-Dock模块,将蛋白晶体结构中天然配体所在位置定义为活性结合口袋.利用Surflex-DockGeomX(SFXC)模块进行高精度分子对接,分子对接参数:配体分子初始构像数目为10,分子片段最大构像数目为20,分子最大可旋转键数据为100,其他参数默认.综合结合构像和对接打分构建αvβ6-多肽复合物用于分子动力学模拟.
本文所有分子动力学模拟均在 Linux工作站用 Amber16[12]计算.αvβ6使用 FF14SB力场,多肽配体使用 GAFF力场和 AM1-BCC电荷,将多肽-蛋白体系浸没于0.15mol/L氯化钠溶液(TIP3P)的立方体盒子中,复合物距立方体盒子边缘为1.00nm,体系包含98个 Na+ ,78个Cl- ,36206个水分子.模拟过程如下:首先进行5步能量优化,避免可能的分子碰撞;其次进行两步加热,使体系温度从0逐渐升温到303.15K,并进行溶液密度调整和体系平衡;最后在303.15K 下对系统(NPT)进行100ns的动力学模拟,时间步长为2fs,压力恒定为1个大气压.结合自由能以及能量分解计算由Amber的 MMPBSA.py程序完成[13],选取最后20ns进行计算并取其平均结构进行结合模式分析,分析αvβ6与配体的结合模式.当结合自由能的值为负值时,体系是稳定的,且该值越小配体-受体结合亲和力越高.
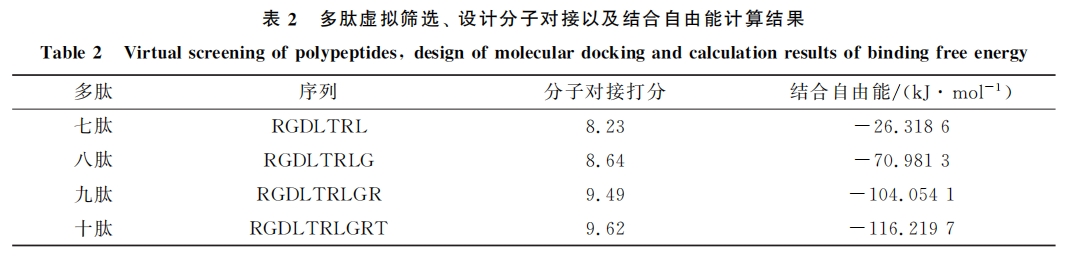
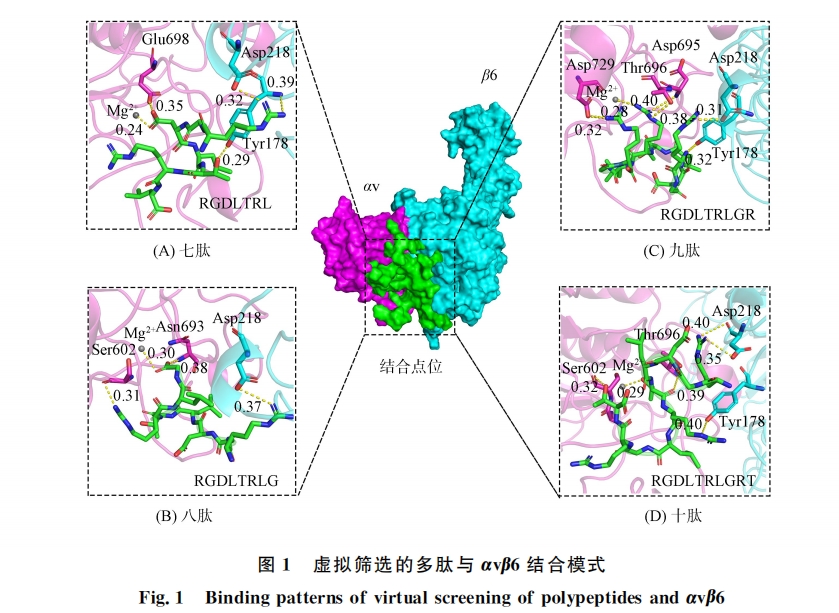
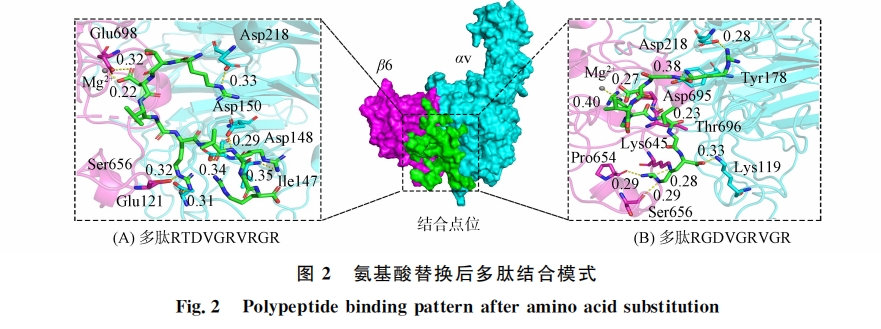
本文虚拟筛选由SYBYL-X1.3完成.首先以 RGDLXXL为基础,在5,6号位通过20种天然氨基酸的随机组合建立一个含400条多肽的多肽文库,通过 Rosetta软件[14]对多肽进行批量结构优化构建虚拟肽库,根据1.2.1节中定义的活性口袋进行虚拟筛选,综合结合构像以及对接打分筛选出最佳结合七肽.然后将筛选出的七肽作为核心逐步延长多肽链,同理筛选出最佳结合八肽、九肽和十肽用于进一步研究.结合模式分析结果表明,现有多肽配体与αvβ6的结合主要为九肽和十肽核心结构,因此将筛选出的九肽、十肽和骨架 RGDLXXL进行氨基酸替换,如亮氨酸可替换为缬氨酸,二者等电点相近,分别为5.98和5.96,侧链结构相似,分别为异丙基和异丁基,二者均为带疏水性侧链的脂肪族氨基酸,氨基酸替换时在改变多肽结构的同时尽量不改变其理化性质.在保持多肽关键结合氨基酸不变的情况下,进一步构建虚拟肽库,通过与上一步相同的虚拟筛选、精准对接、分子动力学模拟获得最佳结合多肽配体,利用分子动力学模拟和结合自由能能量分解计算验证结构改造的合理性.
人源αvβ6蛋白经包被、封闭后,在孔板中加入待测配体溶液.其中空白组仅加入100μLTBS缓冲溶液;空白对照中加入 A20FMDv2-Biotin 和 TBS缓冲溶液各50μL;阳性对照加入 A20Biotin和 A20FMDv2(梯度)各50μL;阴性对照加入 A20FMDv2-Biotin和 A20FMFMDv2-Dv2-GRD各50μL;实验组加入 A20FMDv2-Biotin和待测多肽(梯度)各50Streptavidin-HRP用 PBST 稀 释 100 倍,向 各 孔 加 入μ1L00.每组设置3个平行试验.反应2h后,μL,室 温100μLTMB显色液,立即将孔板置于酶标仪中避光孵育,每5min振 摇 反 应 后,向 各 孔 加 入读取一次650nm 波长处的吸光度(A)值,共读取1h(13个检测点).
2 结果与讨论
根据 高 效 液 相 色 谱 (HPLC)结 果,P1 纯 度 为 98.461%,P2 纯 度 为 95.749%,P3 纯 度 为95.135%,P4纯度为97.554%.根据LC-MS结果,P1计算的摩尔质量为1248.42g/mol,测量的摩尔质量为1249.00g/mol;P2计算的摩尔质量为1196.31g/mol,测量的摩尔质量为1196.70g/mol;P3计算的摩尔质量为1171.31g/mol,测量的摩尔质量为1171.35g/mol;P4计算的摩尔质量为971.07g/mol,测量的摩尔质量为971.25g/mol.可见,经 HPLC以及LC-MS分析,确定合成的多肽为本文研究的目标产物.
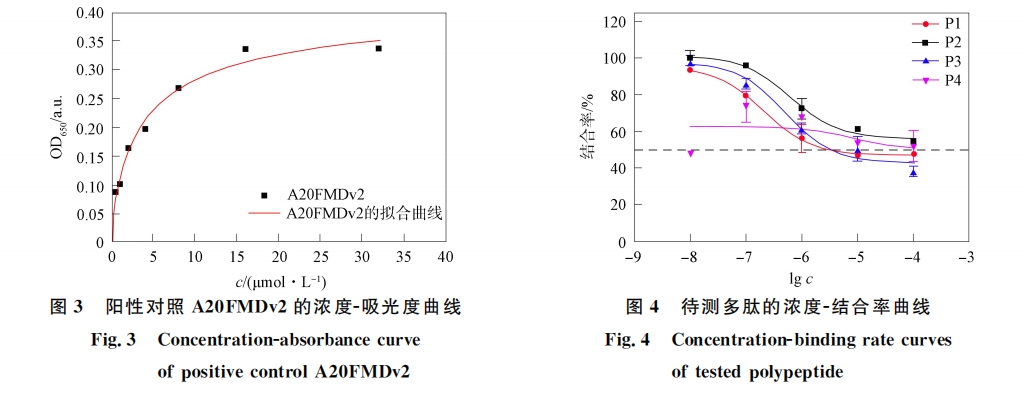
本文通过分子对接与分子动力学模拟分析αvβ6与配体的结合模式,虚拟组合筛选获得符合结合模式的全新结构多肽,采用固相合成法合成多肽,通过间接 ELISA 法测定多肽配体与αvβ6的结合亲和力.实验结果表明:多肽配体 P3与αvβ6具有良好的结合亲和力,略大于阳对照 A20FMDv2的结合亲和力,基本符合设计预期.实验中 P2和 P4由于亲和力较弱,仅存在阳性结果,无法计算其亲和力值.P1虽然也有较强的αvβ6蛋白结合亲和力,但其结构仍具有 RGDLXXL核心,因此在后续的研究中可作为先导化合物改造其结构.P3和 P4虽都为氨基酸替换后筛选出的多肽,但其结合亲和力差距较大.综合分子对接和结合自由能相关计算结果,推测 P4活性较差的原因是其-VGRVGR-部分的空间位阻较小,而αv与β6亚基形成的结合空腔较大,二者结合时存在一定的结合不稳定性.结构生物学研究表明,αvβ6不仅能识别 RGD序列,还能识别-LXXL-基序,该基序折叠成两亲性α-螺旋,结合至β6亚基残基组成的疏水口袋中[15-17].研究中筛选出全新结构多肽含有的-VXXV-序列也可以形成两亲性α-螺旋,该螺旋可与β6亚基形成更多氢键相互作用,RTD 序列也可以与αvβ6形成稳定的氢键,表明了本文采用氨基酸替换方法的合理性.可见,计算机辅助药物设计方法可有效提高药物开发的成功率、降低研发成本并缩短研发周期,是创新药物研发的重要方法.
文献研究表明,整合素αvβ6的表达在许多肿瘤中显著上调,包括口腔鳞癌、乳腺癌、胃癌、胰腺癌、结直肠癌、肺癌等[18-20].整合素αvβ6的表达与许多癌种患者的生存率降低相关,例如结直肠癌、乳腺癌、胰腺导管腺癌、非小细胞肺癌和宫颈鳞状细胞癌等[21-25],并且可以通过纤维连接蛋白和胶原中的 RGD序列参与肿瘤细胞与胞外基质的相互作用,该整合素的过度表达与上皮细胞向间充质样细胞转化有关,可促进肿瘤细胞的侵袭和迁移,通常在肿瘤侵袭病灶的前沿部分中整合素的表达水平较高.目前,靶向整合素成像已成为整合素研究热点之一,包括正电子发射断层扫描和单光子发射计算机断层扫描,整合素显像剂可用于对患者分层进行靶向治疗、评估治疗反应,并监测肿瘤生长[26-30].由于大部分基础生物学仍未完全了解,因此αvβ6为靶标的治疗肿瘤药物也尚未开发,但其仍有作为诊断靶点的潜力.
综上所述,本文采用计算机辅助药物设计的方法设计了一条基于 RTDVXXV 全新结构的αvβ6多肽配体,研究结果表明,该配体具有良好的结合亲和力,配体分子可作为靶向αvβ6多肽或肽类结合物开发的候选分子,为未来αvβ6结合配体的开发提供结构基础.
免责声明:本文为行业交流学习,版权归原作者及原杂志所有,如有侵权,可联系删除。文章标注有作者及文章出处,如需阅读原文及参考文献,可阅读原杂志。